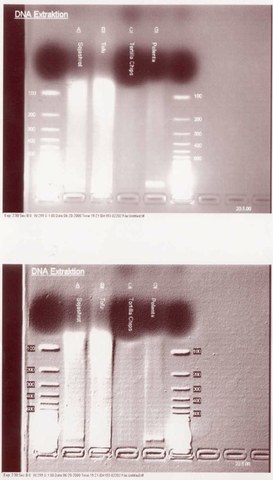
Wir untersuchten Sojaschrot, Bio-Tofu, Tortilla Chips, Polenta
3.1 Gewinnung der DNS
3.1.1. Extraktion der DNS
Die mitgebrachten Lebensmittel wurden zerkleinert und wenn nötig mit Wasser vermengt.
Das homogenisierte Probematerial (Sojaschrot, Bio-Tofu, Tortilla Chips, Polenta ) wird mit Extraktions-Puffer, Guanidin-hydrochlorid und Proteinase gemischt.
Der Extraktions-Puffer schützt die Desoxiribonukleinsäuren, die stark auf pH-Schwankungen reagieren und ohne die Reaktionszusätze von den im Homogenat vorhandenen Nukleasen aufgelöst würden. Guanidin-hydrochlorid reagiert mit Enzymen, die die DNS angreifen. Die Proteinase löst alle Proteine auf, somit auch den Zellkern und die Verpackung der DNS.
3.1.2. Reinigung der DNS
Mittels Wizard Harz werden die DNS Stränge zusammen gehalten. Dadurch bleiben sie in einem Filter als Rückstand. Die DNS wird mit Isopropanol gewaschen.
Das Harz wir durch heisses deionisiertes Wasser aufgelöst.
Die reine DNS Lösung wird mit 14000 G (Erdbeschleunigung) zentrifugiert.
3.2.2. Mastermix
Der Mastermix dient als Kontrolle für die anderen Proben, ob überhaupt DNS vorhanden ist.
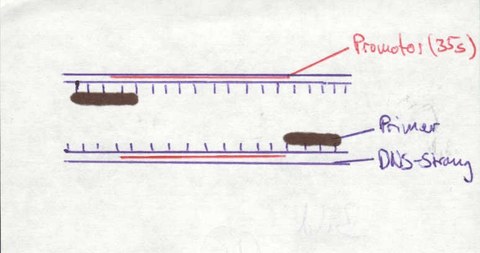
3.2.3. Auswertung des Nachweises des 35S-Promotors
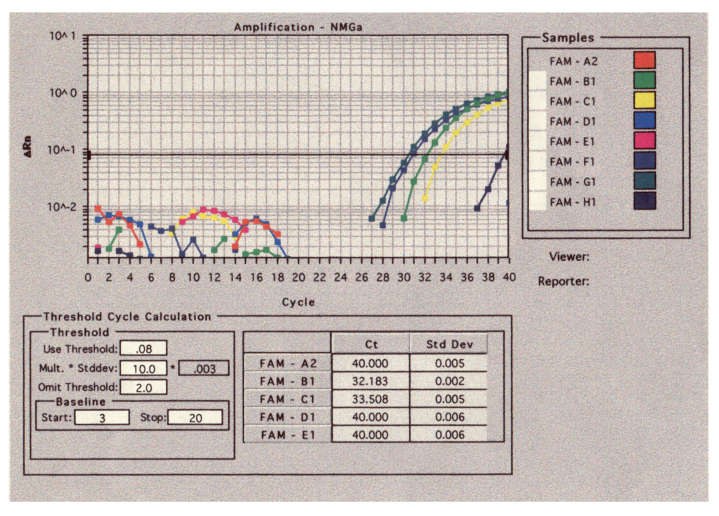
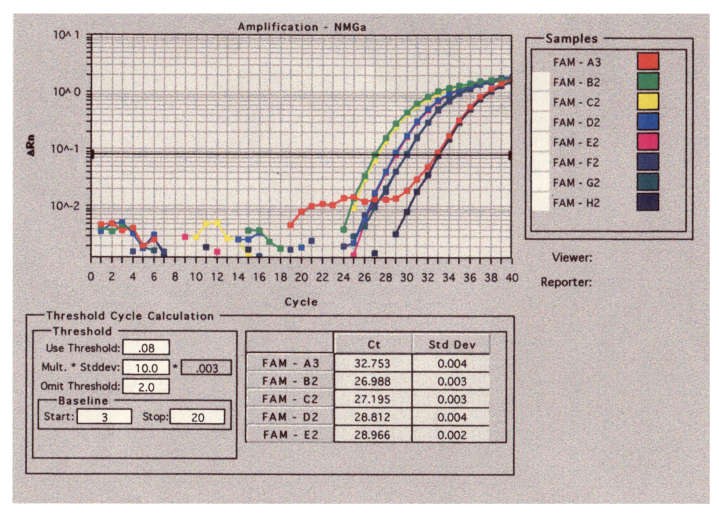
Die Länge des Fragmentes wird durch Vergleich mit einem DNS-Längenstandard abgeschätzt. Wenn ein Fragment von 195 Basenpaaren Länge vorhanden ist, dann ist in der untersuchten DNS-Lösung 35S-Promotor-DNS vorhanden.
3.3 Elektrophorese
Mit der Agarose-Gelelektrophorese werden DNS-Stücke entsprechend ihrer Länge aufgetrennt. Es lassen sich Nukleinsäure-Fragmente im Grössenbereich von 30 bis 10`000 Basenpaaren auftrennen und dokumentieren.
3.3.1 Herstellung des Agarose-Gels
Wir stellten mit TBE-Pufferstammlösung, deionisiertem Wasser und Agarose (je nach Basenpaarenlänge eine entsprechende Konzentration) ein Gemisch her, das wir aufkochen damit sich die Agarose auflöst.
3.3.5 Die negativ geladene DNS wandert
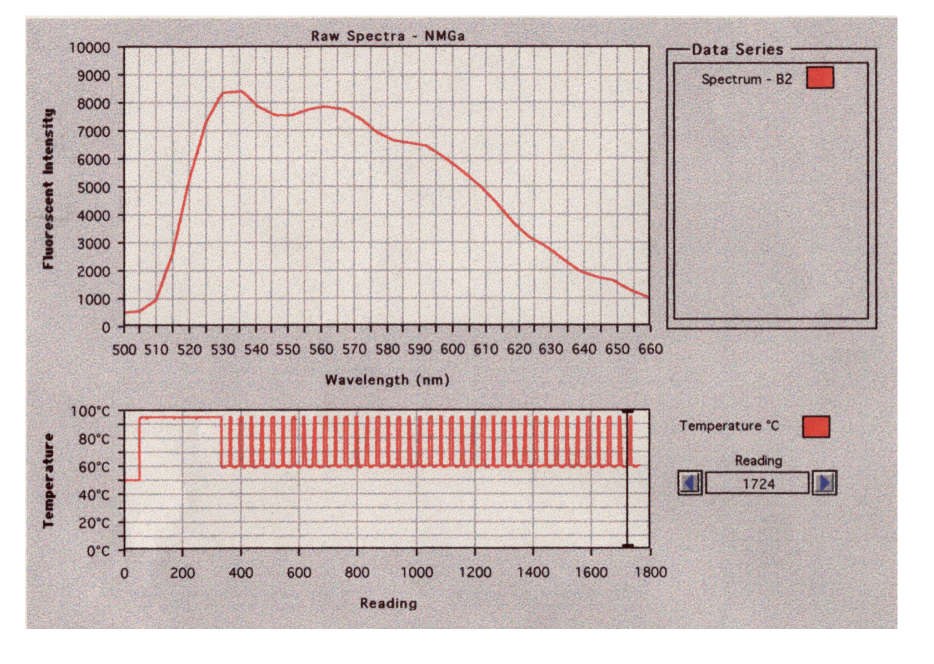
Die negativ geladene DNS wandert durch das Anlegen einer Gleichspannung zur Anode. Die DNS-Fragmente werden durch das Molekularsieb des Agarose-Gels nach ihrer Grösse separiert. Die DNS wird mit dem fluoreszierenden Farbstoff Ethidiumbromid (das stark kanzerogen ist) angefärbt und im UV-Licht sichtbar gemacht.
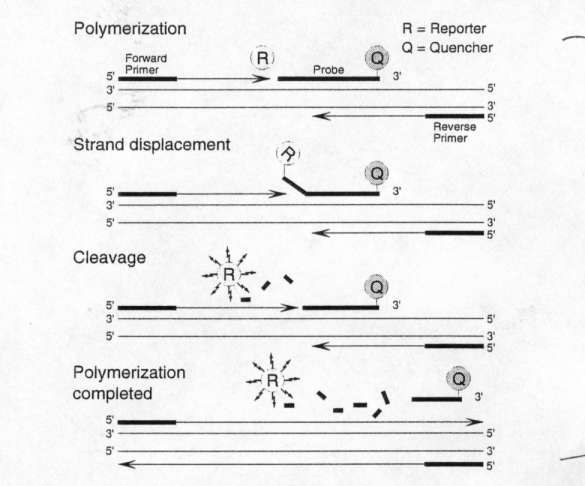
3.4 Der Taq-Man
Idee:
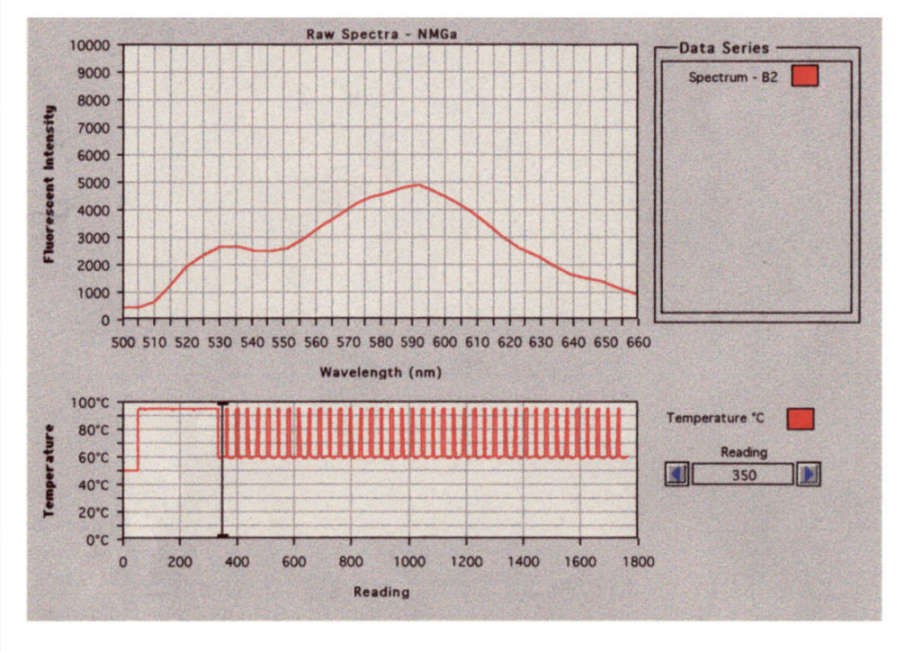
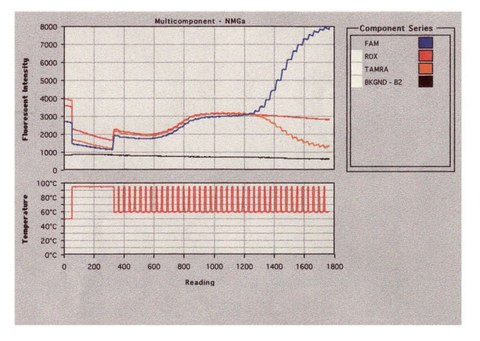
(GA)Der Taq Man ist eine realtime Analyse einer PCR. Die Analyse basiert auf der Fluoreszenz einzelner Moleküle, die sich an die DNS heften und während der PCR abgelöst werden.
Prinzip:
Es werden sog. Sonden in die Nährlösung gegeben. Diese enthalten zwei optisch aktive Gruppen an deren Ende. Wird eines der beiden Enden aktiviert, strahlt nur eines mit einer bestimmten Frequenz zurück. Das bedeutet, das die optisch aktiven Gruppen einzeln fluoreszieren können, nahe beieinander aber nur das eine strahlt.
Was hat das mit DNS-Analyse zu tun?
Durch die PCR werden die DNS-Stränge aufgespaltn. Die Fluoreszenzmittel können sich anbinden. Nur eines der Enden fluoresziert. Durch die fortschreitende PCR wird das zweite Ende abgeschnitten.